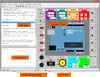
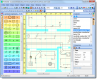
TinkerCell es una herramienta de software de diseño asistido por computadora para la biología sintética. Combina la interfaz visual con la API de programación (Python, Octave, C, Ruby) y permite a los usuarios compartir su código entre sí a través de un repositorio central.
TinkerCell es una herramienta de diseño asistido por computadora (CAD) para biología sintética. Dado que la biología sintética es un campo en rápida evolución, estos son los problemas que se tuvieron en cuenta al diseñar TinkerCell:
- las técnicas de modelado en biología aún se encuentran en una etapa de desarrollo
- Las técnicas experimentales evolucionan constantemente, especialmente los métodos de automatización y evolución dirigida.
- las bases de datos de componentes biológicos aún están madurando
En resumen, TinkerCell está diseñado con la anticipación de que el futuro de la biología sintética será una interacción intrincada entre una variedad de técnicas experimentales, bases de datos que almacenan resultados de experimentos y modelos matemáticos que explican diferentes aspectos de los experimentos.
El caso de uso del programa CAD ideal sería el siguiente:
- Paso 1: el usuario dibuja un sistema biológico
- Paso 2: el usuario realiza algunos análisis
- Paso 3: regrese al paso 1 si el análisis no es satisfactorio
El paso de análisis puede incluir potencialmente:
- análisis matemático de sistemas no lineales
- simulaciones estocásticas, análisis estructural y otros métodos de biología de sistemas
- predicción de trayectorias evolutivas para evolución dirigida
- Análisis y optimización de la secuencia de ADN.
- búsqueda en la base de datos para encontrar componentes adecuados
- generar diferentes versiones del mismo diseño
- cargar datos experimentales de bases de datos y relacionarlos con el diagrama
- ...más...
Es demasiado tener todas estas funciones en TinkerCell desde el principio. Por lo tanto, TinkerCell utiliza un marco de plug-in flexible, que permitirá que otros contribuyan con nuevas funciones a TinkerCell. Los complementos se pueden escribir en C, C ++, Python u Octave. Otros idiomas, p. Java, también podría agregarse a esta lista.
La idea básica detrás de TinkerCell es representar un diagrama que sea lo suficientemente detallado para que pueda mapearse a modelos o resultados experimentales. Un investigador humano puede conectar modelos y experimentos porque puede vincularlos utilizando una comprensión conceptual del sistema. TinkerCell habilita esa capacidad en software.
No es del todo correcto proporcionar un solo modelo matemático para un sistema biológico porque un modelo matemático es solo una hipótesis. Puede haber múltiples hipótesis para el mismo sistema. TinkerCell distingue un "modelo" de un "diagrama".
Si bien los diagramas de TinkerCell son muy abstractos, también contienen todos los detalles necesarios para los diferentes tipos de análisis. Un diagrama no es un modelo en sí mismo, pero tiene suficiente información para generar uno o más modelos. Por ejemplo, aquí hay un diagrama abstracto.
TinkerCell es modular, lo que significa que cada proceso en el diagrama anterior puede consistir en subprocesos. En el diagrama de ejemplo anterior, el proceso de catálisis enzimática tiene procesos dentro de él. Los procesos internos se pueden cambiar sin cambiar el diagrama externo. La siguiente ilustración muestra cómo los subprocesos se asignan al diagrama principal mediante la identificación de los "tipos" de cada componente.
Metas a largo plazo
El objetivo a largo plazo de TinkerCell es convertirse en una de las aplicaciones clave involucradas en la ingeniería eficiente de sistemas biológicos (celulares). La forma en que esto funcionará es conectando TinkerCell a las herramientas de automatización de laboratorio. En esta situación, los usuarios dibujarían un sistema en TinkerCell. Algún complemento de TinkerCell enviaría la información a un robot remoto. Cuando regresen los resultados de los experimentos, otro complemento de TinkerCell recomendaría automáticamente los modelos a utilizar en función de la evidencia experimental. Para habilitar esta canalización, es importante representar que el modelo inicial es un formato genérico pero detallado; las herramientas de software existentes no pueden hacer esto, razón por la cual una de las muchas razones por las cuales la tubería no es posible actualmente.
Capacidades de análisis actuales
- simulación determinista (usando COPASI)
- simulación estocástica (usando COPASI)
- cálculo de estado estacionario (usando COPASI)
- computación jacobiana (usando COPASI)
- computación de valor propio (usando COPASI)
- simulación estocástica exacta (usando COPASI)
- simulación estocástica basada en salto tau (usando COPASI)
- simulación híbrida (usando COPASI)
- análisis estructural de la matriz de estequiometría (usando libStructural)
- análisis de supuestos del modelo, como los supuestos de equilibrio
- exploración de parámetros (Monte Carlo y análisis de estado estacionario)